PythonでPDFから表を抽出してcsvに変換する(Bing AIにサポートしてもらいながら)
背景
農林水産省が提供している「各種抗生物質・合成抗菌剤・駆虫剤・抗原虫剤の販売高と販売量」という資料がある。
この資料、1ページ目にタイトル、2ページ目から最終ページまで表、という形式になっているのだが、2019年分まではPDFでしか提供されていない。(2020年分からはExcelファイルも提供されている。)
コピペしてテキスト整形しようにもテキストの位置がガタガタで上手くいかない。Excelの「データの取得」でPDFを開くのを試しても、ページ毎に別々のシートに分割されるし(これはまあいい)、ヘッダー行・見出し列がなぜかシート毎に異なる行・列に配置されるし、セル内改行があると改行位置で行分割されるし、で「整形に何時間かかるんだ…」なファイルにしかならない。ということでRでなんとかできないかとtabulizerパッケージを試してみたのだがこれまた上手くいかず、PDF内で複数列に分かれているデータが同じ列として認識されてしまう。
ということで、最近Pythonを勉強しているのと生成系AIとの付き合い方を模索中なのもあり、Bing AIにPythonコードを書かせてみた。
実践
プロンプト
次のPDFについて話をしましょう。 https://www.maff.go.jp/nval/iyakutou/hanbaidaka/pdf/h17hanbaikoukin20180205.pdf このPDFに含まれる表をcsv化する方法を探しています。 以下の手順で、Python用のコードを作成してください。 - Pythonのcamelotライブラリからread_pdf()を使用し、2ページ目から最終ページまでの表を読み取る。その際line_scaleは適切なものを使用する。 - 読み取られた表のリストに含まれる全ての表から1~2行目を削除する。 - 表のリストを縦に結合して1つの表を作成する。 - 列名は以下を使用する:class,group,substance,administration,sales,amount,beef,dairy,horse,pig,broiler,layer,dogcat,freshwater,seawater,ornamental,others - substance列に「成分・計」の文字が含まれる行は削除する。 - group列に「系統名・計」の文字が含まれる行は削除する。 - セル内に含まれる改行は半角スペースに置換する。 - sales列からothers列までは数値形式に変換する。 - beef列からothers列に含まれる空白セルには代わりに0を入力する。 - 作成した表をcsv形式で保存する。
ライブラリと使用する関数の選定は自分で行った。
回答されたコード
import camelot import pandas as pd # PDFから表を読み取る tables = camelot.read_pdf('https://www.maff.go.jp/nval/iyakutou/hanbaidaka/pdf/h17hanbaikoukin20180205.pdf', pages='2-end', line_scale=40) # 全ての表から1~2行目を削除し、表のリストを縦に結合 df = pd.concat([table.df.iloc[2:] for table in tables]) # 列名を設定 df.columns = ['class', 'group', 'substance', 'administration', 'sales', 'amount', 'beef', 'dairy', 'horse', 'pig', 'broiler', 'layer', 'dogcat', 'freshwater', 'seawater', 'ornamental', 'others'] # substance列に「成分・計」の文字が含まれる行とgroup列に「系統名・計」の文字が含まれる行を削除 df = df[~df['substance'].str.contains('成分・計')] df = df[~df['group'].str.contains('系統名・計')] # セル内に含まれる改行を半角スペースに置換 df.replace('\n', ' ', regex=True, inplace=True) # sales列からothers列までを数値形式に変換 cols = ['sales', 'amount', 'beef', 'dairy', 'horse', 'pig', 'broiler', 'layer', 'dogcat', 'freshwater', 'seawater', 'ornamental', 'others'] df[cols] = df[cols].apply(pd.to_numeric, errors='coerce') # beef列からothers列に含まれる空白セルには代わりに0を入力 df[cols] = df[cols].fillna(0) # 作成した表をcsv形式で保存 df.to_csv('output.csv', index=False)
結果
このコードを試すと無事csv化できた。
ちなみに思い通りの結果が得られるまでに3度ほどリテイクを出している。
感想
実はBing AIにコードを書かせる前に自分でPythonコードを書いていたしそれで成功もしていた。しかしふと思いついてBing AIにコードを書かせてみると自分で書くより数倍速かった。
まあPythonに慣れたらこれぐらいの内容は自分でコードを書く方が速いんだろうが、初心者のうちは生成系AIにコードを書いてもらって、それを自分で写経しつつエラーを修正していくのはかなり有用かもしれない。
みんなの自動翻訳@TexTraのWebAPIをRから使用する
目的
みんなの自動翻訳@TexTra® の質問・要望欄を見ていたら、Web APIのRでのアクセス例の追加要望があったのでスクレイピングの練習も兼ねてやってみた。
コード
汚いコードですが動くのでOKです。
余力があったらパッケージ化してGitHubに上げる上げた。 (GitHub - fmsan51/minhon)
if (!require("pacman")) install.packages("pacman"); library(pacman) pacman::p_load(httr, rvest) # ユーザーID login_id <- "User ID here" # https://mt-auto-minhon-mlt.ucri.jgn-x.jp/content/mt/ から適当なものを選択 # 以下の例は英→日 api_key <- "API key here" api_secret <- "API secret here" base_url <- "https://mt-auto-minhon-mlt.ucri.jgn-x.jp" api_name <- "mt" api_param <- "generalNT_en_ja" # 翻訳対象テキスト text <- "The quick brown fox jumps over the lazy dog" endpoint <- oauth_endpoint(NULL, NULL, paste0(base_url, "/oauth2/token.php")) app <- oauth_app("minhon", api_key, api_secret) token <- oauth2.0_token(endpoint, app, client_credentials = T) res <- POST(paste0(base_url, "/api/"), body = list(access_token = token$credentials$access_token, key = api_key, api_name = api_name, api_param = api_param, name = login_id, type = "xml", # jsonも可 text = text)) res_text <- html_element(content(res, encoding = "UTF-8"), xpath = "/resultset/result/information/text-t") html_text(res_text)
RStudioのEast Asian Width問題をなんとかしたい(願望)
※ただの作業メモ。
目的未達。
誰かやってください...。
目的
RStudioで○や△や※を入力するとカーソル位置がおかしくなる問題(East Asian Width問題)をなんとかしたい。

参考
背景
従来のRStudioはQtWebEngineを用いて作成されていた。こちらのコードペイン部分はQTermWidgetを使用していると思われる。そしてEast Asian Width問題に関連すると思われるissueがこちら。
2016年2月にopenされ、2021年2月以降動きがない。
2022年6月、RStudio PreviewのElectron版がリリースされた。また今後RStudioはQtWebEngineからElectronに移行する予定だそうだ。
RStudio Electron Preview Release Notes – RStudio Support
Electron版のコードペイン部分はAceを使用していると思われる。East Asian Width問題に関連すると思われるissueがこちら。
2021年10月にopenされ、関連するisssueは多いが修正の動きはない。
まあ正直QTermWidget以上に動きはないのだが、コードをちらちら読んでいたら「自分でもなんとかなるかも」と思ったので途中まで作業してみたメモ。
作業内容
カーソル表示位置に関連するコードはおそらくこの部分。
ace/text.js at 3f934510514a25c53edf64bb80911a96b7133908 · ajaxorg/ace
re = ...を修正してその直後のwhileループ部分に場合分けを追加してやればなんとかなる気がする。
Unicodeの EastAsianWidth.txt (version 14.0.0) から作成したregexが以下。
[\u00A1\u00A4\u00A7-\u00A8\u00AA\u00AD-\u00AE\u00B0-\u00B4\u00B6-\u00BA\u00BC-\u00BF\u00C6\u00D0\u00D7-\u00D8\u00DE-\u00E1\u00E6\u00E8-\u00EA\u00EC-\u00ED\u00F0\u00F2-\u00F3\u00F7-\u00FA\u00FC\u00FE\u0101\u0111\u0113\u011B\u0126-\u0127\u012B\u0131-\u0133\u0138\u013F-\u0142\u0144\u0148-\u014B\u014D\u0152-\u0153\u0166-\u0167\u016B\u01CE\u01D0\u01D2\u01D4\u01D6\u01D8\u01DA\u01DC\u0251\u0261\u02C4\u02C7\u02C9-\u02CB\u02CD\u02D0\u02D8-\u02DB\u02DD\u02DF\u0300-\u036F\u0391-\u03A9\u03B1-\u03C1\u03C3-\u03C9\u0401\u0410-\u044F\u0451\u2010\u2013-\u2016\u2018-\u2019\u201C-\u201D\u2020-\u2022\u2024-\u2027\u2030\u2032-\u2033\u2035\u203B\u203E\u2074\u207F\u2081-\u2084\u20AC\u2103\u2105\u2109\u2113\u2116\u2121-\u2122\u2126\u212B\u2153-\u2154\u215B-\u215E\u2160-\u216B\u2170-\u2179\u2189\u2190-\u2199\u21B8-\u21B9\u21D2\u21D4\u21E7\u2200\u2202-\u2203\u2207-\u2208\u220B\u220F\u2211\u2215\u221A\u221D-\u2220\u2223\u2225\u2227-\u222C\u222E\u2234-\u2237\u223C-\u223D\u2248\u224C\u2252\u2260-\u2261\u2264-\u2267\u226A-\u226B\u226E-\u226F\u2282-\u2283\u2286-\u2287\u2295\u2299\u22A5\u22BF\u2312\u2460-\u24E9\u24EB-\u254B\u2550-\u2573\u2580-\u258F\u2592-\u2595\u25A0-\u25A1\u25A3-\u25A9\u25B2-\u25B3\u25B6-\u25B7\u25BC-\u25BD\u25C0-\u25C1\u25C6-\u25C8\u25CB\u25CE-\u25D1\u25E2-\u25E5\u25EF\u2605-\u2606\u2609\u260E-\u260F\u261C\u261E\u2640\u2642\u2660-\u2661\u2663-\u2665\u2667-\u266A\u266C-\u266D\u266F\u269E-\u269F\u26BF\u26C6-\u26CD\u26CF-\u26D3\u26D5-\u26E1\u26E3\u26E8-\u26E9\u26EB-\u26F1\u26F4\u26F6-\u26F9\u26FB-\u26FC\u26FE-\u26FF\u273D\u2776-\u277F\u2B56-\u2B59\u3248-\u324F\uE000-\uF8FF\uFE00-\uFE0F\uFFFD\u1F100-\u1F10A\u1F110-\u1F12D\u1F130-\u1F169\u1F170-\u1F18D\u1F18F-\u1F190\u1F19B-\u1F1AC\uE0100-\u10FFFD]
作業ここまで。
誰かやってくれないかなー。
追記
github.com
これを参考に修正していけばなんとかなる.....だろうけど多すぎてやる気なくなってきたな......。
group_by() + summarise() でgroupごとにベクトルを結合する
目的
library(tidyverse) # これを data <- tribble(~x, ~y, "a", c(1, 2), "a", c(), "a", c(3), "b", c(), "c", c(4), "c", c(5) ) # こうしたい tribble(~x, ~y, "a", c(1, 2, 3), "b", c(), "c", c(4, 5) )
これが簡単そうにみえて意外と手こずったのでメモ。
結論
list(do.call(c, y))
試行錯誤録
失敗
data |> group_by(x) |> summarise(y = c(y)) #> `summarise()` has grouped output by 'x'. You can override using the `.groups` #> argument. #> # A tibble: 6 × 2 #> # Groups: x [3] #> x y #> <chr> <list> #> 1 a <dbl [2]> #> 2 a <NULL> #> 3 a <dbl [1]> #> 4 b <NULL> #> 5 c <dbl [1]> #> 6 c <dbl [1]>
普通にc()で結合しようとしてもうまくいかない。
面倒くさい方法
data |> group_by(x) |> summarise(y = list(y)) |> mutate(y = map(y, flatten_dbl)) #> # A tibble: 3 × 2 #> x y #> <chr> <list> #> 1 a <dbl [3]> #> 2 b <dbl [0]> #> 3 c <dbl [2]>
できてるっちゃできてるんだけど一見「なんでわざわざlist()した後にflatten()してるの?」と思うし、わざわざmutate()せずにsummarise()だけでできる方法がある気がする。
成功
data |> group_by(x) |> summarise(y = list(do.call(c, y))) #> # A tibble: 3 × 2 #> x y #> <chr> <list> #> 1 a <dbl [3]> #> 2 b <NULL> #> 3 c <dbl [2]>
これでOK。
ggplot2のヒートマップで中間値を任意に指定する
目的
ggplot2でヒートマップを作るときに中間値1を任意に指定したい。
最終的に記事下部のやり方その2に着地。
例
このプロットで0を青、1を黄色、2000を赤にする。
library(ggplot2) data <- faithfuld data$z <- faithfuld$density * 50000 ggplot(data, aes(waiting, eruptions, fill = z)) + geom_tile() + scale_fill_gradient2(low = "blue", mid = "yellow", high = "red")

プロット中にも凡例にも青が見えない。
失敗例
最初はmidpointとlimitを設定してやればいいだろ......と思っていたのだが実際やってみると以下の通り。青が見えない。
ggplot(data, aes(waiting, eruptions, fill = z)) + geom_tile() + scale_fill_gradient2(low = "blue", mid = "yellow", high = "red", midpoint = 1, limits = c(0, 2000))

成功例
その1:values = rescale(c(最小値, 中間値, 最大値))
やり方その1。scale_fill_gradientn()にvalues = rescale(c(最小値, 中間値, 最大値))を設定する。
ggplot(data, aes(waiting, eruptions, fill = z)) + geom_tile() + scale_fill_gradientn(colours = c("blue", "yellow", "red"), values = rescale(c(0, 1, 2000)))

問題点
- 青の部分がべったりしているのが気になる(対処可能:やり方2参照)。
- legendに青が見えない。
その2:breaks/labels/limits = c(最小値, 中間値, 最大値)
やり方その2。scale_fill_gradientn()のbreaks・labels・limitsにc(最小値, 中間値, 最大値)を設定する。
ggplot(data, aes(waiting, eruptions, fill = z)) + geom_tile() + scale_fill_gradientn(colours = c("blue", "yellow", "red"), na.value = "blue", breaks = c(1 / 2000, 1, 2000), labels = c(0, 1, 2000), limits = c(1 / 2000, 2000), trans = "log")
今回は1をlegendの中央に持ってくるためにtrans = "log"を設定しているので最小値を1/2000にしているが、log変換しないのならば最小値は0でいい。
na.value = "最小値の色"の指定もlog変換しないなら不要。データ中にNAが存在する場合は、na.valueを指定する代わりにプロット前に最小値未満の値を最小値に置換しておく。

legendに青が見えるようになり、値が分かりやすくなった。
問題点?
- やはり青がべったりしている。
coloursとbreaksの指定を増やすことで対処できるはず(試していない)だが今回は面倒くさいのでそこまでしていない。 - legendの0の横が凹んでいる(分かりづらいだけで実は1の横も凹んでいる)。
guide_colorbar()のticksまわりの設定で対処可能のはず(試していない)。
以上。
-
中央値ではない ↩
Rmarkdown+TinyTeXでBIZ UDP明朝を使う
目的
ユニバーサルデザインフォントが好きだ。
RmarkdownのKnit to PDFで作成されるPDFにもUDフォントを使いたい。
ということでRmarkdown+TinyTeX環境でBIZ UDP明朝を使用するためのYAMLヘッダ設定。
追記(2022/06/14)
Quarto が完成すると Jupyter でも R Markdown のように簡単にスライドや文書を作れるかもしれない
こちらの記事を読んで、XeLaTeXを使用していたのをLuaLaTeXに書き直した。
YAMLヘッダ(LuaLaTeX版)
output: pdf_document: latex_engine: lualatex extra_dependencies: luatexja-preset: ["bizud", "bold"] documentclass: bxjsarticle classoption: - pandoc - ja=pandoc
※(一切使用しないのだが)原ノ味フォントのインストール(tinytex::tlmgr_install("haranoaji"))が必要。
インストールしていないとコンパイル時に
\JY3/mc/m/n/10=file:HaranoAjiMincho-Regular.otf:-kern;jfm=ujis at 9.24713pt not loadable: metric data not found or bad.
というエラーが出る。
XeLaTeX版より記載がだいぶすっきりした。
難点としてはXeLaTeXより処理が遅い気がする。
LuaLaTeXの方が日本語の扱い方がいいらしい?のでLuaLaTeXでいくことにする。
Quarto版追記(2022/07/29)
format: pdf: pdf-engine: lualatex documentclass: bxjsarticle luatexja-preset: ["bizud", "bold"] classoption: - pandoc - ja=pandoc
YAMLヘッダ(XeLaTeX版)
output: pdf_document: latex_engine: xelatex header-includes: | \usepackage{zxjatype} \setCJKmonofont[BoldFont=BIZ UDGothic Bold]{BIZ UDMincho Medium} mainfont: BIZ UDPMincho Medium mainfontoptions: - Scale=MatchUppercase - BoldFont=BIZ UDPGothic Bold CJKmainfont: BIZ UDPMincho Medium CJKoptions: - Scale=1 - BoldFont=BIZ UDPGothic Bold
BIZ UDP明朝にはBoldフォントが用意されていないのでBIZ UDPゴシック Boldで代用。
明朝体のBoldはRegularと見分けづらいため元々好きではなく、太字にする際にはゴシック体のBoldを使用しているため無問題。
ほんとはmainfontもゴシック体を使用したいが学術的な内容のPDF文章をゴシック体で書くと違和感が強いかも、ということで仕方なく明朝体を採用。
斜体はない。諦めろ。
結果

参考
2022/06/14追加:
Rで生存時間分析
そういえば生存時間分析を自分でしたことがなかったので、この間読んだ論文のデータを使って試してみた。
データ
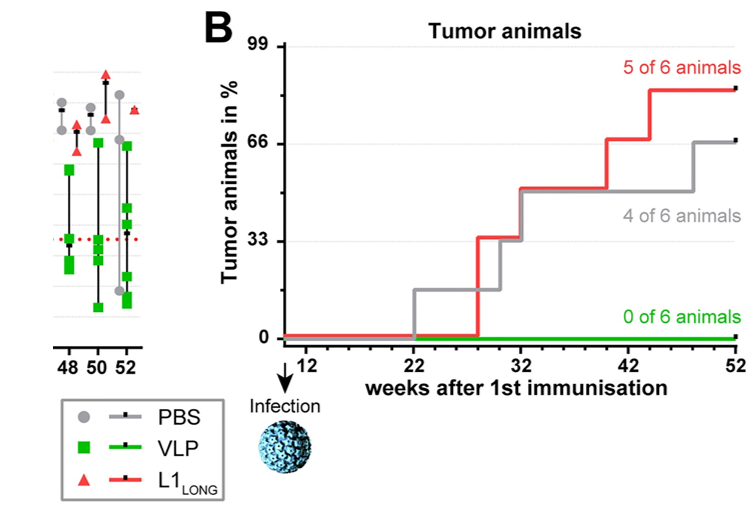
Hasche D, Ahmels M, Braspenning-Wesch I, Stephan S, Cao R, Schmidt G, Müller M and Rösl F (2022) Isoforms of the Papillomavirus Major Capsid Protein Differ in Their Ability to Block Viral Spread and Tumor Formation. Front. Immunol. 13:811094. doi: 10.3389/fimmu.2022.811094
(CC BY 4.0)
より Figure 5B のデータを使用。
コード
library(tibble) # tribble用 library(dplyr) # mutate用 library(survival) library(survminer)
## Loading required package: ggplot2
## Loading required package: ggpubr
library(broom) # glance(p-valueの算出)用
dat <- tribble( ~vaccination, ~weeks, ~censored, "PBS", 22L, 1L, "PBS", 30L, 1L, "PBS", 32L, 1L, "PBS", 48L, 1L, "PBS", 52L, 0L, "PBS", 52L, 0L, "VLP", 52L, 0L, "VLP", 52L, 0L, "VLP", 52L, 0L, "VLP", 52L, 0L, "VLP", 52L, 0L, "VLP", 52L, 0L, "L1long", 28L, 1L, "L1long", 28L, 1L, "L1long", 32L, 1L, "L1long", 40L, 1L, "L1long", 44L, 1L, "L1long", 52L, 0L ) |> mutate(vaccination = factor(vaccination, c("PBS", "VLP", "L1long"))) dat |> group_by(vaccination) |> summarise(life = median(weeks))
## # A tibble: 3 × 2
## vaccination life
## <fct> <dbl>
## 1 PBS 40
## 2 VLP 52
## 3 L1long 36
論文中に記載されている各群の生存期間(median)と一致するためデータの再現はOKだと思われる。
Surv(dat$weeks, dat$censored)
## [1] 22 30 32 48 52+ 52+ 52+ 52+ 52+ 52+ 52+ 52+ 28 28 32 40 44 52+
sf <- survfit(Surv(weeks, censored) ~ vaccination, data = dat) ggsurvplot(sf)
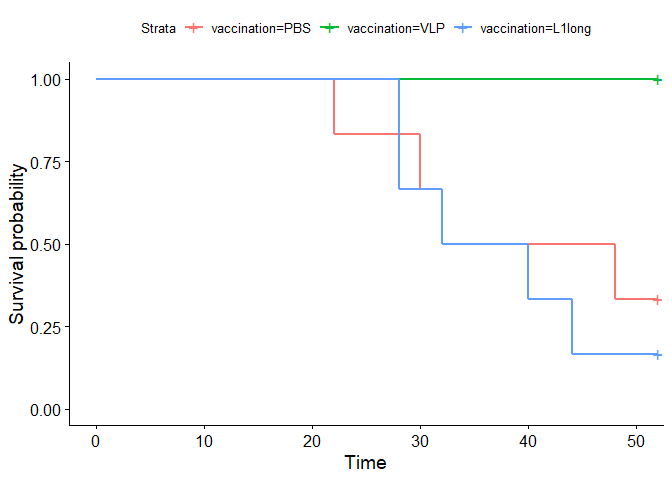
プロット。いい感じ。
set.seed(1) ggsurvplot(sf, fun = "event", xlim = c(10, 52), ylim = c(-0.02, 1.02), position = position_jitter(h = 0.02), censor = F)
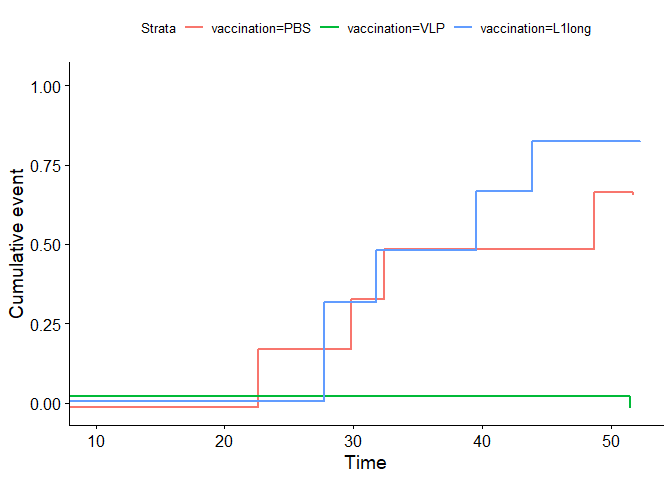
より論文中のFigure 5Bに近づけるとこんな感じ?
survdiff(Surv(weeks, censored, origin = 10) ~ vaccination, data = dat, subset = vaccination %in% c("PBS", "VLP")) |> glance()
## # A tibble: 1 × 3
## statistic df p.value
## <dbl> <dbl> <dbl>
## 1 5.57 1 0.0183
survdiff(Surv(weeks, censored, origin = 10) ~ vaccination, data = dat, subset = vaccination %in% c("VLP", "L1long")) |> glance()
## # A tibble: 1 × 3
## statistic df p.value
## <dbl> <dbl> <dbl>
## 1 8.24 1 0.00410
origin = 10なのはこの論文中でInfectionが10週目に行われるため。
結果は論文中に記載のp値と一致する。
多重性の補正は? と思わなくもない。Dunnettの補正だと0.05/2=0.025でどちらも有意。