Rで生存時間分析
そういえば生存時間分析を自分でしたことがなかったので、この間読んだ論文のデータを使って試してみた。
データ
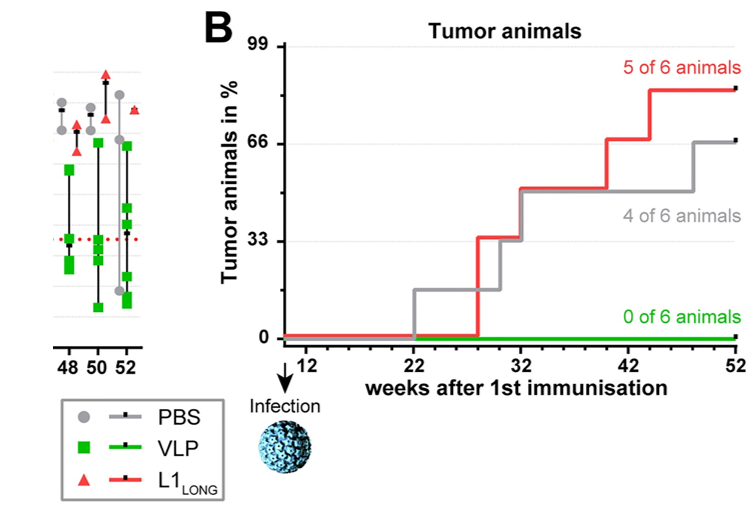
Hasche D, Ahmels M, Braspenning-Wesch I, Stephan S, Cao R, Schmidt G, Müller M and Rösl F (2022) Isoforms of the Papillomavirus Major Capsid Protein Differ in Their Ability to Block Viral Spread and Tumor Formation. Front. Immunol. 13:811094. doi: 10.3389/fimmu.2022.811094
(CC BY 4.0)
より Figure 5B のデータを使用。
コード
library(tibble) # tribble用 library(dplyr) # mutate用 library(survival) library(survminer)
## Loading required package: ggplot2
## Loading required package: ggpubr
library(broom) # glance(p-valueの算出)用
dat <- tribble( ~vaccination, ~weeks, ~censored, "PBS", 22L, 1L, "PBS", 30L, 1L, "PBS", 32L, 1L, "PBS", 48L, 1L, "PBS", 52L, 0L, "PBS", 52L, 0L, "VLP", 52L, 0L, "VLP", 52L, 0L, "VLP", 52L, 0L, "VLP", 52L, 0L, "VLP", 52L, 0L, "VLP", 52L, 0L, "L1long", 28L, 1L, "L1long", 28L, 1L, "L1long", 32L, 1L, "L1long", 40L, 1L, "L1long", 44L, 1L, "L1long", 52L, 0L ) |> mutate(vaccination = factor(vaccination, c("PBS", "VLP", "L1long"))) dat |> group_by(vaccination) |> summarise(life = median(weeks))
## # A tibble: 3 × 2
## vaccination life
## <fct> <dbl>
## 1 PBS 40
## 2 VLP 52
## 3 L1long 36
論文中に記載されている各群の生存期間(median)と一致するためデータの再現はOKだと思われる。
Surv(dat$weeks, dat$censored)
## [1] 22 30 32 48 52+ 52+ 52+ 52+ 52+ 52+ 52+ 52+ 28 28 32 40 44 52+
sf <- survfit(Surv(weeks, censored) ~ vaccination, data = dat) ggsurvplot(sf)
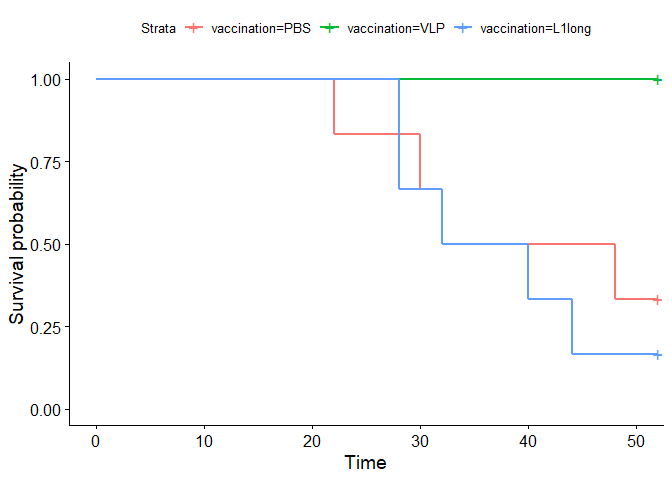
プロット。いい感じ。
set.seed(1) ggsurvplot(sf, fun = "event", xlim = c(10, 52), ylim = c(-0.02, 1.02), position = position_jitter(h = 0.02), censor = F)
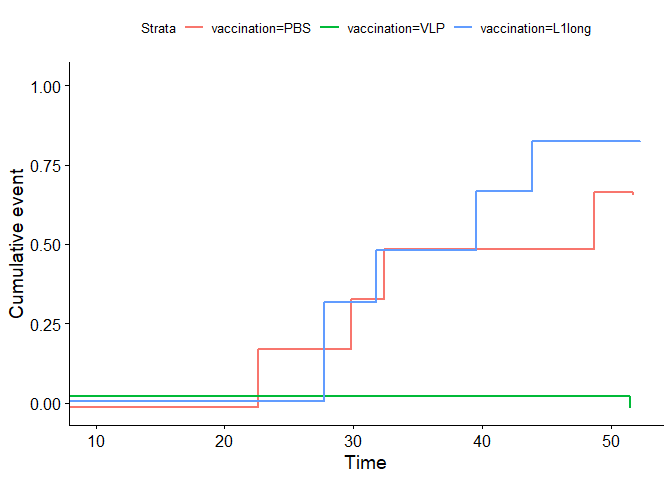
より論文中のFigure 5Bに近づけるとこんな感じ?
survdiff(Surv(weeks, censored, origin = 10) ~ vaccination, data = dat, subset = vaccination %in% c("PBS", "VLP")) |> glance()
## # A tibble: 1 × 3
## statistic df p.value
## <dbl> <dbl> <dbl>
## 1 5.57 1 0.0183
survdiff(Surv(weeks, censored, origin = 10) ~ vaccination, data = dat, subset = vaccination %in% c("VLP", "L1long")) |> glance()
## # A tibble: 1 × 3
## statistic df p.value
## <dbl> <dbl> <dbl>
## 1 8.24 1 0.00410
origin = 10なのはこの論文中でInfectionが10週目に行われるため。
結果は論文中に記載のp値と一致する。
多重性の補正は? と思わなくもない。Dunnettの補正だと0.05/2=0.025でどちらも有意。